Kladistik
Die Kladistik (altgriechisch κλάδος klados „Zweig“) oder phylogenetische Systematik ist eine Methodik der biologischen Systematik und Taxonomie auf der Basis der Evolutionsbiologie.
Sie wurde von dem deutschen Entomologen Willi Hennig in den 1950er Jahren in ihren Grundzügen umrissen und in seinem Lehrbuch Phylogenetic Systematics 1966 beschrieben.
Der Ausdruck „Kladistik“ für diese Methode, die auf der Verwendung von Kladen genannten geschlossenen Abstammungsgemeinschaften beruht, wurde ursprünglich von dem Evolutionsbiologen Ernst Mayr eingeführt, der damit die von Hennig selbst „phylogenetische Systematik“ genannte Methodik aber nicht neutral beschreiben, sondern kritisieren wollte.[1][2] Insbesondere im deutschen Sprachraum hatte daher der Ausdruck Kladistik lange Zeit einen negativen Beiklang und wurde von den Befürwortern der Methode gemieden, Hennig selbst verwendete ihn etwa nur in Anführungszeichen.[3] Durch die Verwendung von cladistics im englischen Sprachraum ohne diese wertende Färbung wird er heute meist ohne wertenden Beiklang gebraucht. So gibt die Willi Hennig Society ihre Zeitschrift unter dem Titel Cladistics heraus.[4]
Die kladistische Methodik, auf der Basis von Apomorphien schematische Darstellungen (Diagramme) phylogenetischer Verwandtschaftsbeziehungen, sogenannte Kladogramme, als Grundlage der biologischen Systematik zu konstruieren, ist heute allgemein akzeptierter Standard in der Biologie; alternative Konzepte wie die Phänetik sind nur noch von historischem Interesse. Die Kladistik auch als ausschließliche taxonomische Methode zu verwenden, ist zwar überwiegender wissenschaftlicher Standard, wird aber von einer Gruppe Biologen bis heute kritisiert und von ihnen selbst nicht praktiziert.[5]
Zielsetzung
[Bearbeiten | Quelltext bearbeiten]Die phylogenetische Systematik bezweckt, ein System der Organismen zu erstellen, das ausschließlich auf phylogenetischer Verwandtschaft basiert. Da tatsächlich, wie von der Evolutionstheorie überzeugend begründet, alle Organismen miteinander verwandt sind (Deszendenztheorie), gibt es dabei nur ein korrektes System, das die tatsächlich auf der Erde abgelaufene Evolution abbilden würde. Da aber diese natürliche Verwandtschaft auf Vorgängen beruht, die Jahrmillionen in der Vergangenheit liegen, kann sie nicht direkt beobachtet, sondern nur aus Indizien erschlossen werden. Die Aufgabe der Systematik besteht darin, dieses natürliche System zu finden. Neue taxonomische Gruppen, wie zum Beispiel neue Arten, sind dabei dadurch entstanden, dass schon bestehende Arten sich aufgespalten haben, indem verschiedene Populationen innerhalb einer Art sich auseinanderentwickelt haben (vgl. Artbildung). Alle höheren taxonomischen Einheiten (Gruppen wie die Wirbeltiere, die Säugetiere, die Blütenpflanzen) gehen dabei letztlich auch ursprünglich einmal auf einzelne Stammarten zurück, so dass für die höheren Einheiten (Makroevolution) kein anderes System benötigt wird als für die tieferen (Mikroevolution).[6]
Gruppen innerhalb eines auf Verwandtschaft basierenden Systems sind jedoch nicht immer leicht erkennbar, wenn die Organismen nur nach ihrer Ähnlichkeit zueinander klassifiziert werden. Eine Stammgruppe kann sich in zwei Gruppen von Nachkommen evolutiv aufspalten, von denen eine ihren Ahnen relativ ähnlich sieht, während eine andere radikal anders aussehen kann. Beispielsweise bilden die Vorfahren der heute lebenden Quastenflosser, der Lungenfische und der Landwirbeltiere eine gemeinsame Verwandtschaftsgruppe, deren Vorfahr einem Quastenflosser weitaus ähnlicher sah als einem Menschen oder einem Elefanten. Zudem ähnelt der Quastenflosser in der generellen Körpergestalt anderen Fischen weitaus stärker als den Landwirbeltieren, mit denen er näher verwandt ist als mit diesen. Da beide von gemeinsamen (ebenfalls fischähnlichen) Vorfahren abstammen, sind sie natürlich auch, wenn auch etwas entfernter, miteinander verwandt. Das heißt: Solche Gruppen sind nicht immer monophyletisch. Die Gruppe der Fische beispielsweise basiert auf phylogenetischer Verwandtschaft, aber trotzdem sind die Fische keine monophyletische Gruppe (Fische enthalten als Nachfahren alle anderen Wirbeltiere).
Eine Kladistik ist eine spezielle phylogenetische Systematik, bei der alle Gruppen auch monophyletisch sind. Eine monophyletische Gruppe (auch Klade genannt) enthält alle Nachfahren einer Stammart sowie die Stammart selbst, jedoch keine Arten, die nicht Nachfahren dieser Stammart sind. Die Merkmalsausstattung der Stammart entspricht dem während der Analyse zu rekonstruierenden Grundmuster. Grundlage für die Erstellung monophyletischer Gruppen sind gemeinsame abgeleitete Merkmale, so genannte Synapomorphien. Das Grundmuster repräsentiert die Gesamtheit der nicht abgeleiteten Merkmale (Plesiomorphien) der Gruppen. Im Gegensatz zum idealen Bauplan, welcher die Gesamtheit aller Merkmale einer Gruppe in sich vereint, entspricht also der Grundplan dem Körperbau und der Merkmalsausprägung einer Art, die real existiert hat.
Das Ergebnis einer kladistischen Analyse ist eine Verwandtschaftshypothese, die als Kladogramm dargestellt wird. Hennig bezeichnet das Kladogramm als „Argumentationsschema der phylogenetischen Systematik“. Anders als ein Stammbaum hat das Kladogramm nur terminale Taxa. Es lässt damit also nicht die Entwicklung einer rezenten Form aus einer anderen zu, oder, anders ausgedrückt, keine rezente (lebende) Art kann und darf Stammart einer anderen rezenten Art sein. Fossile Arten können in ein Kladogramm integriert werden, sie bilden dann aber ebenfalls terminale Taxa. Das bedeutet, die Zuordnung einer fossilen Art als der tatsächlichen Stammart wird vermieden. Knoten eines Kladogramms stellen die Stammart der beiden aus ihr hervorgehenden Schwestergruppen dar. Autapomorphien der jeweiligen Schwestergruppen sind abgeleitete Merkmale, die allen Taxa dieser Gruppe gemeinsam sind und die bei der Schwestergruppe nicht auftreten.
Phylogenetische Systematik ist eine historische Wissenschaft, da man die Phylogenese der Organismen nicht beobachten, sondern nur rekonstruieren kann. Daher werden alle Verwandtschaftshypothesen immer nicht experimentell zu bestätigende Hypothesen bleiben. Die phylogenetische Systematik versucht, widerspruchsfreie Hypothesen aufzustellen und Verwandtschaftshypothesen, die miteinander in Konflikt stehen, aufzulösen. Die Methodik der phylogenetischen Systematik gibt dem Wissenschaftler ein Instrumentarium an die Hand, das es ihm erlaubt, seine Argumentation reproduzierbar darzulegen.
Kladogramme
[Bearbeiten | Quelltext bearbeiten]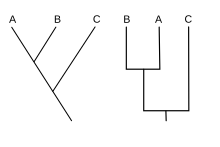
Die Darstellung der Verwandtschaftsverhältnisse erfolgt in so genannten Kladogrammen. Diese unterscheiden sich von evolutionären Stammbäumen in den folgenden Punkten:
- Bei einer Verzweigung gibt es immer nur zwei Äste (dichotome Verzweigung).
- Die Verzweigungen werden nicht gewichtet, man hat also kein Maß für die Änderung, um es in einem Kladogramm darzustellen. (In evolutionären Stammbäumen kann man ein solches Maß in unterschiedlichen Streckenlängen für Abzweigungen darstellen, siehe auch Divergenz).
- Es gibt keine absolute Zeitachse.
- Alle Artspaltungsereignisse werden so realistisch wie möglich dargestellt.
Jeder Ast ist durch ein abgeleitetes Merkmal begründet. Was dieses Merkmal jeweils sein soll, ist Gegenstand der Forschung. So kann man zum Beispiel Plazentatiere über ihre Plazenta von den Beuteltieren unterscheiden, diese wiederum besitzen gegenüber den Plazentatieren z. B. eine Reduktion der ausgebildeten Milchzähne. Der namensgebende Beutel ist allerdings keine Synapomorphie, sondern ist innerhalb der Beuteltiere mehrfach entstanden (Konvergenz), auch besitzen nicht alle Beuteltiere einen Beutel bzw. einige haben ihn reduziert.
Kladogramm der Säugetiere
[Bearbeiten | Quelltext bearbeiten]Dieser Zusammenhang soll hier am Beispiel eines vereinfachten Kladogramms der Säugetiere dargestellt werden:
| Säugetiere |
| ||||||||||||
Wichtig ist, dass alle Äste mindestens eine Autapomorphie aufweisen.
Merkmale des Grundmusters können innerhalb der Gruppe wieder verloren gehen. Dies ist dann eine Autapomorphie des betroffenen Taxons. Ein Beispiel hierfür ist der sekundäre Verlust der Flügel bei vielen Fluginsekten (Pterygota).
Kladogramm „Mensch, Gorilla und Schimpanse“
[Bearbeiten | Quelltext bearbeiten]Charles Darwin nahm an, dass zwischen den unten aufgeführten Arten die nächste Verwandtschaft zwischen Gorillas und Schimpansen bestehe und der Mensch eine Sonderstellung habe. Stephen Jay Gould sah Indizien dafür, dass Menschen und Schimpansen sich am nächsten stehen und sich die Gorillas in der Entwicklungsgeschichte früher abgespalten haben.
| |||||||||||||||||||
Kladogramm nach Mark Abraham
Topologie
[Bearbeiten | Quelltext bearbeiten]Grundsätzlich kann ein Stammbaum auf unterschiedliche Weise dargestellt werden, indem bei einer Verzweigung (oder gleich mehreren) einzelne Zweige miteinander vertauscht werden (mathematisch eine Permutation). In obigen Kladogrammen könnte man z. B. Menschen und Schimpansen (oder Theria und Monotremata) miteinander vertauschen. Trotz unterschiedlichen Aussehens stellten die Kladogramme jeweils denselben Sachverhalt, genannt Topologie, dar. Eine jeweils andere Topologie läge vor, wenn auf der Abstammungslinie zum Menschen zuerst die Schimpansen und erst später die Gorillas abzweigten.
Konstruktion von Kladogrammen
[Bearbeiten | Quelltext bearbeiten]Bei einer kladistischen Analyse einer Gruppe von Taxa wird versucht, deren Verwandtschaftsverhältnisse zu rekonstruieren und das resultierende Verzweigungsmuster als Kladogramm abzubilden. Ausgangspunkt der Analyse ist es in der Regel, so viele aussagekräftige Merkmale wie nur möglich zu sammeln. Möglicherweise aussagekräftig sind dabei alle Merkmale, die bei einem Teil der analysierten Taxa vorkommen, bei einem anderen Teil nicht. Entgegen dem früheren Vorgehen in der Systematik verzichtet man heute in der Regel darauf, die Merkmale zu gewichten. Gibt man einigen Merkmalen a priori ein höheres Gewicht als anderen, besteht die Gefahr, dass Vorurteile des Bearbeiters die Analyse verzerren. Nützlich für die Analyse sind dabei ausschließlich Merkmale, die Synapomorphien darstellen. Gemeinsam ererbte Stammgruppenmerkmale (Symplesiomorphien) sind für die Analyse irrelevant, (Aut-)Apomorphien eines einzelnen Taxons betonen zwar dessen Eigenständigkeit, sind aber für seine Verwandtschaft nicht erhellend. Normalerweise werden die berücksichtigten Merkmale in Form einer Charaktermatrix aufbereitet und dargestellt, in der die Ausprägung des Merkmals durch eine Zahl kodiert wird (z. B. „Flügel vorhanden“ = 1, „flügellos“ = 0).
Für die Analyse muss selbstverständlich vorher (anhand der Remaneschen Homologiekriterien) die Homologie der Merkmale so weit wie nur möglich geklärt worden sein. Gemeinsame Merkmale, die konvergent entstanden sind, oder Merkmale, die auf paralleler Evolution beruhen (das sind Merkmale, die bei nah verwandten Arten auf vergleichbarer genetischer Grundlage unabhängig voneinander entstanden sind), können die Analyse verzerren, wenn sie unerkannt bleiben. Sind betrachtete Merkmale durch Konvergenzen und zahlreiche unabhängige Entstehungen und Rückbildungen so geprägt, dass sie kaum noch zur Analyse beitragen, spricht man von Homoplasie. In vielen Fällen ist es unmöglich, den Grad der Homoplasie vorher zu bestimmen. Starke Homoplasie der analysierten Merkmale entwertet das resultierende Kladogramm, weshalb zur Homoplasie neigende Merkmale so weit wie möglich vermieden werden sollten.
Das Hauptproblem bei der kladistischen Analyse sind Rückbildungen von Merkmalen. Betrachtet man z. B. die Gruppen der Felsenspringer (Archaeognatha), Eintagsfliegen (Ephemeroptera) und Flöhe (Siphonaptera) anhand des Merkmals „Flügelausbildung“, so sind Flügel nur bei den Eintagsfliegen vorhanden. Das Fehlen der Flügel liegt im Fall der Felsenspringer daran, dass sie von primär ungeflügelten Vorfahren abstammen. Die Flöhe hingegen hatten geflügelte Vorfahren, nur sind bei ihnen die Flügel ohne jeden Rest zurückgebildet worden. Sind in diesem Fall die Zusammenhänge aufgrund zahlreicher anderer Merkmale noch eindeutig aufklärbar, ist dies bei zahlreichen anderen Gruppen keinesfalls mit gleicher Sicherheit möglich. Es kommt sogar vor, dass einzelne Stammlinien ein Merkmal zurückbilden und es dann bei einzelnen Linien der Nachkommen unabhängig voneinander ein zweites Mal erworben wird. Die Frage, ob ein bestimmtes Merkmal bei der Stammgruppe vorhanden war und also bei den Linien, die es nun nicht aufweisen, rückgebildet worden ist oder ob es bei den Vorfahren fehlte und innerhalb der betrachteten Gruppe in einer oder mehreren Linien neu erworben wurde, wird die „Polarität“ des Merkmals genannt. Zur Bestimmung der Polarität werden sogenannte Außengruppen in die Analyse mit einbezogen. Eine Außengruppe kann jedes Taxon sein, das mit den analysierten Arten zwar verwandt ist, das aber mit Sicherheit und klar außerhalb des betrachteten Verwandtschaftskreises steht. Aus naheliegenden Gründen sollte es ein wenig spezialisiertes Taxon mit wenigen Autapomorphien sein. Die Wahl der Außengruppe(n) kann die Analyse erheblich beeinflussen.
In der klassischen Vorgehensweise Hennigs wurde ein Kladogramm nun dadurch konstruiert, dass man durch Klärung der Polaritäten eine hypothetische Stammform konstruierte und dann die betrachteten Taxa durch einzelnes oder gruppenweises Hinzufügen ausprobierend so lange anordnete, bis sich ein überzeugender Stammbaum ergab. In der modernen kladistischen Analyse wird dieser Schritt von einem Sortieralgorithmus übernommen. Dazu werden die durch die Merkmalsmatrix beschriebenen Taxa so lange permutiert, bis ein Verzweigungsmuster minimaler Länge gefunden ist. Die Außengruppen-Taxa können in diese Analyse mit einbezogen werden. Dieses kürzeste Verzweigungsmuster gilt dann als wahrscheinlichste Hypothese der Verwandtschaftsverhältnisse. Dies wird als parsimony (engl. „Geiz“, „Sparsamkeit“) bezeichnet. Am häufigsten verwendet wird das Sortierprogramm PAUP (Phylogenetic Analysis Using Parsimony), es gibt allerdings eine Reihe weiterer gängiger Programme. Gibt es zwei oder mehr unterschiedliche Stammbäume gleicher Länge, sind dies gleichwertige Hypothesen, und die Verwandtschaft ist nicht entscheidbar. Erstes Analyseergebnis ist allerdings einfach ein Verbindungsgraph. Um diesen in ein Kladogramm zu verwandeln, muss die Polarität geklärt sein (d. h., es muss klar sein, welche der Verzweigungen zuerst stattgefunden haben). Dies kann entweder aus der Datenanalyse abgeleitet werden oder (bei guter Kenntnis der Außengruppen) erzwungen werden. Dieser Schritt wird als „Verwurzelung“ (engl. rooting) des Kladogramms bezeichnet.
Biologische Systematik
[Bearbeiten | Quelltext bearbeiten]Die biologische Systematik versteht sich heute als eine Wissenschaft, die Lebewesen anhand ihrer Abstammung klassifiziert. Daher ist die Kladistik eine ihrer Arbeitsmethoden.
Bei der Erstellung eines Kladogramms werden Eigenschaften der betrachteten Lebewesen verglichen. Es werden oft, aber nicht ausschließlich, morphologische Merkmale, Charakteristika des Stoffwechsels und genetische Informationen benutzt.
Danach wird eine Vielzahl von Kladogrammen erstellt. Dasjenige Kladogramm mit der geringsten Anzahl von notwendigen Veränderungen innerhalb des angenommenen Evolutionsverlaufes gilt als das wahrscheinlichste. Oft ist es bei der Angabe eines Kladogramms von Interesse, andere Kladogramme, die mit einer sehr ähnlichen Anzahl von Veränderungen konstruiert sind, ebenfalls zu betrachten.
Die Bioinformatik bedient sich für die Rekonstruktion von Kladogrammen diverser Standardsoftware, die multiple Sequenzalignments und die Variabilität einzelner Reste auswerten, wie zum Beispiel Phyogeny Inference Package (PHYLIP).[7]
Die traditionelle Namensgebung in der Biologie kann die baumartige Struktur der evolutionären Entwicklung nicht fassen. Daher wird eine phylogenetische Namensgebung, PhyloCode genannt, diskutiert.
Kladistischer Status von Taxa
[Bearbeiten | Quelltext bearbeiten]Nicht immer lassen sich die traditionell gebräuchlichen Einteilungseinheiten (Taxa) der biologischen Klassifikation oberhalb des Ranges einer Art wegen der ihnen jeweils zugrundeliegenden Konzepte problemlos in eine kladistisch basierte, moderne Systematik übernehmen. Zudem können durch Nutzung molekularer statt morphologischer Daten in der Biologie oder durch neue Fossilfunde und damit eine Erweiterung der bestehenden Datenbasis in der Paläontologie neue Verwandtschaftsanalysen bereits untersuchter Gruppen zu neuen Ergebnissen führen, mit Konsequenzen für vormals definierte Taxa. So werden für Taxa oberhalb des Ranges einer Art folgende kladistische Status unterschieden:
Monophyletisch
[Bearbeiten | Quelltext bearbeiten]
Das Taxon hat eine jüngste gemeinsame Stammform (engl. most recent common ancestor, MRCA) und umfasst auch alle Untergruppen, die sich von dieser Stammform herleiten, sowie die Stammform selbst, jedoch keine anderen Gruppen. Das Monophylum begründet sich durch Apomorphien der gemeinsamen Stammform und wird auch als geschlossen bezeichnet.
- Beispiel: Die Metazoa umfassen alle tierischen Mehrzeller. Diese haben das apomorphe Merkmal Mehrzelligkeit gemeinsam.
Eine alternative Bezeichnung für ein Monophylum ist Klade. In der modernen Systematik ist prinzipiell jedes Taxon immer auch ein Monophylum. Traditionelle Taxa, die sich als monophyletisch erweisen, sind auch in der modernen Systematik allgemein anerkannt.
Paraphyletisch
[Bearbeiten | Quelltext bearbeiten]
Das Taxon hat zwar eine jüngste gemeinsame Stammform, enthält aber nicht alle Untergruppen, die auf diese Stammform zurückgehen, wie es beim Monophylum der Fall ist. Ein Paraphylum gründet sich auf die Symplesiomorphien der enthaltenen Taxa und wird auch als offen bezeichnet.
- Beispiel: Die Reptilien sind paraphyletisch, da die Vögel klassischerweise nicht zu ihnen gezählt werden, obwohl deren letzte gemeinsame Stammart ein Dinosaurier war und sie somit denselben Stamm haben wie alle anderen Tierarten der Gruppe der Reptilien. Das Taxon der Sauropsida, welches die Klasse der Reptilien und die Klasse der Vögel zusammenfasst, ist hingegen monophyletisch.
Traditionelle Taxa, die sich als paraphyletisch erweisen, erfahren in der Regel entweder nur noch informell Verwendung oder werden als Monophyla neu definiert, woraufhin sie dann auch jene Gruppen enthalten, die sie als Monophylum enthalten müssen, traditionell jedoch nicht enthielten. Im Englischen wird ein Paraphylum auch als „grade“ bezeichnet, als Gegenstück zum „clade“, dem Monophylum.
Polyphyletisch
[Bearbeiten | Quelltext bearbeiten]
Das Taxon hat keine gemeinsame Stammform, die jünger ist als die gemeinsamen Stammformen, die seine Untertaxa mit anderen Taxa haben.
- Beispiel: Die Würmer („Vermes“) umfassen verwandtschaftlich weit voneinander entfernte Gruppen. Weitere Beispiele sind die warmblütigen Tiere („Endothermia“), Wasservögel, Süßwasserfische und Bäume.
Solche Taxa sind jedoch oft schon vor vielen Jahrzehnten als „unnatürliche Gruppierungen“ erkannt worden, sodass sie heute allgemein nicht als systematische Begriffe Verwendung finden.
Literatur
[Bearbeiten | Quelltext bearbeiten]- Peter Ax: Das phylogenetische System. Systematisierung der lebenden Natur aufgrund ihrer Phylogenese. Fischer, Stuttgart u. a. 1984, ISBN 3-437-30450-X.
- Peter Ax: Systematik in der Biologie. Darstellung der stammesgeschichtlichen Ordnung in der lebenden Natur (= UTB 1502). Fischer, Stuttgart 1988, ISBN 3-437-20419-X.
- T. Ryan Gregory: Understanding Evolutionary Trees. In: Evolution: Education and Outreach. Bd. 1, Nr. 2, 2008, S. 121–137, doi:10.1007/s12052-008-0035-x (Volltext frei zugänglich).
- Willi Hennig: Grundzüge einer Theorie der phylogenetischen Systematik. Deutscher Zentralverlag, Berlin 1950.
- Willi Hennig: Phylogenetische Systematik (= Pareys Studientexte. Nr. 34). Paul Parey, Berlin u. a. 1982, ISBN 3-489-60934-4.
- Willi Hennig: Aufgaben und Probleme stammesgeschichtlicher Forschung (= Pareys Studientexte. Nr. 35). Paul Parey, Berlin u. a. 1984, ISBN 3-489-61534-4.
- Olivier Rieppel: Einführung in die computergestützte Kladistik. Dr. Friedrich Pfeil, München 1998, ISBN 3-931516-57-1.
- Walter Sudhaus, Klaus Rehfeld: Einführung in die Phylogenetik und Systematik. Gustav Fischer, Stuttgart u. a. 1992, ISBN 3-437-20475-0.
- Johann Wolfgang Wägele: Grundlagen der Phylogenetischen Systematik. Dr. Friedrich Pfeil, München 2000, ISBN 3-931516-73-3.
- Bernhard Wiesemüller, Hartmut Rothe, Winfried Henke: Phylogenetische Systematik. Eine Einführung. Springer, Berlin u. a. 2003, ISBN 3-540-43643-X.
- Rainer Willmann: Die Art in Raum und Zeit. Das Artkonzept in der Biologie und Paläontologie. Paul Parey, Berlin u. a. 2005, ISBN 3-489-62134-4.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- OneZoom: Tree of Life – Fraktaler, zoombarer Stammbaum aller rezenten Lebewesen-Arten (optional auch Polytomie-Ansicht)
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Ernst Mayr: Principles of Systematic Zoology. McGraw-Hill, 1969.
- ↑ Ernst Mayr: Cladistic analysis or cladistic classification? In: Zeitschrift für zoologische Systematik und Evolutionsforschung 12 (1974), S. 94–128.
- ↑ Willi Hennig: Kritische Bemerkungen zur Frage Cladistic Analysis or Cladistic Classification. In: Zeitschrift für zoologische Systematik und Evolutionsforschung 12 (1974), S. 279–294; englische Übersetzung durch C.D. Griffiths: Cladistic Analysis or Cladistic Classification? A Reply to Ernst Mayr. In: Systematic Zoology, 24 (1975), S. 244–256.
- ↑ Willi Hennig Society. Abgerufen am 29. März 2016.
- ↑ phylogenetische Systematik – Lexikon der Biologie. In: spektrum.de. Abgerufen am 29. März 2016.
- ↑ Edward O. Wiley, Bruce S. Lieberman: Phylogenetics. Theory and Practice of Phylogenetic Systematics. 2. Auflage. A John Wiley & Sons, 2011, ISBN 978-0-470-90596-8.
- ↑ Bernard R. Baum: PHYLIP: phylogeny inference package. version 3.2 by Joel Felsenstein. In: The Quarterly Review of Biology. Band 64, Nr. 4, Dezember 1989, S. 539–541, doi:10.1086/416571.